2. Lösung des Traveling Salesman Problem mit dem Genetischen Algorithmus¶
Der in diesem Abschnitt beschriebene Python Code ist im Modul geneticAlgo.py zusammengefasst. Die Implementierung ist keine typische Python-Implementierung. Der rein prozedurale Ansatz begründet sich darin, dass ich den Algorithmus ursprünglich in Matlab geschrieben hatte und das Matlab Script mit relativ wenig Aufwand in ein Python/Numpy Programm übertragen habe. Das Programm implemenitert die Lösung des Travelling Salesman Problem (TSP) mit dem Genetischen Algorithmus (GA). Die einzelnen Schritte des GA sind nicht in allgemeiner Form implementiert, sondern speziell für die Lösung des TSP.
2.1. Das Travelling Salesman Problem¶
Ein Handlungsreisender muss eine Menge von Städten besuchen und zum Schluss wieder in die Stadt, in der er seine Reise begann, zurück kehren. Es ist davon auszugehen, dass zwischen allen Städten direkte Verbindungen bestehen. Das Handlungsreisende (Traveling Salesman) steht vor dem Problem die Reihenfolge der zu besuchenden Städte derart festzulegen, dass die geamte Distanz seiner Reise minimal ist. Die Lösung des TSP ist NP-complete. Eine suboptimale Lösung kann z.B. mit dem GA berechnet werden.
2.2. Allgemeiner Ablauf des Genetischen Algorithmus¶

2.3. Die Teilschritte des Genetischen Algorithmus in der Anwendung TSP-Lösung¶
2.3.1. Darstellung der Chromosomen¶
Im Fall des TSP wird ein Chromosom repräsentiert durch eine Rundreise, welche alle Städte passiert und in der Stadt endet in welcher sie begann. Die Population besteht dann aus einer Anzahl (POPSIZE) solcher Rundreisen. Eine Rundreise wird dann als ein Vektor mit 11 Elementen modelliert. Jedes Element ist ein Integer zwischen 1 und 10. Ausser der Zahl, welche die Ausgangsstadt repräsentiert, darf jeder Integer im Vektor nur einmal vorkommen. Das elfte Element ist immer gleich dem ersten Element. Eine mögliche Rundreise wäre also [ 3 6 5 2 8 7 9 4 1 10 3 ].
2.3.2. Fitness¶
Für das TSP bietet sich eine Berechnung der Fitness in Abhängigkeit von der Gesamtdistanz einer Rundreise an. Allerdings ist die direkte Benutzung der Distanz nicht angebracht, denn per Definition soll der Fitness Wert proportional zur Eignung (Nutzen), d.h. antiproportional zu den Kosten eines Chromosoms sein. Für die Kosten eines Chromosoms wird die Gesamtdistanz, für die Fitness der inverse Kostenwert (1/Kosten) verwendet.
2.3.3. Selektion¶
Die Auswahl eines Chromosomenpaars aus der Population, soll derart implementiert werden, dass die Wahrscheinlichkeit für die Auswahl eines Chromosomes umso größer ist, je höher sein Fitness-Wert ist. Realisiert werden kann dies indem eine in den Grenzen zwischen 0 und Summe über alle Fitness-Werte gleichverteilte Zufallszahl erzeugt wird. Der ganze Wertebereich der Zufallszahl wird in Abschnitte unterteilt, deren Größe genau den Fitnesswerten der einzelnen Chromosomen entspricht. Der Bereich in welchen die gewürfelte Zufallszahl fällt bestimmt das Chromosom das selektiert wird.
2.3.4. Kreuzung¶
Für die Kreuzung wird zunächst ein zufälliger Kreuzungspunkt innerhalb des Vektors (eines ausgewählten Chromosomes) gewürfelt. Für das TSP wird folgende Realisierung des Crossover gewählt. Der Anfang (Head) des neuen Vektors ist bis zum Kreuzungspunkt indentisch mit dem Anfang des alten Vektors. Der zweite Teil vom Kreuzungspunkt bis zum Ende des Vektors (Tail), wird wie folgt bestimmt. Scanne vom Anfang bis zum Ende des zweiten ausgewählten Vektors. Sobald dort eine Zahl gefunden wird, die noch nicht im neu zu bildenden Chromosom steht, füge diese Zahl an die nächste freie Stelle im Tail des neu zu bildenden Chromosoms ein. An der letzten Stelle des neuen Chromosomes muss die gleiche Zahl stehen wie an seiner ersten Stelle. Dieser Prozess wird für beide ausgewählte Chromosomen durchgeführt. Die Wahrscheinlichkeit mit der eine Kreuzung in einer Iteration ausgeführt sollte als Parameter einstellbar sein (Typisch: Wahrscheinlichkeitswert nahe bei 1).
Beispiel:
Die Kreuzung der beiden Chromosomen
- [ 3 6 5 2 8 7 9 4 1 10 3 ]
- [ 7 8 10 2 1 3 6 9 4 5 7 ]
am Kreuzungspunkt cp=6 ergibt die beiden Kinder
- [ 3 6 5 2 8 7 9 10 1 4 3 ]
- [ 7 8 10 2 1 3 6 5 9 4 7 ]
2.3.5. Mutation¶
Mit einer eher kleinen Wahrscheinlichkeit wird in einer Iteration auch eine Mutation der ausgewählten und evtl. durch Kreuzung modifizierten Chromosomen durchgeführt. Im Fall des TSP wird Mutation wie folgt implementiert. Falls eine Mutation stattfindet wird ein zufälliges Paar von Indizees mit Werten zwischen 1 und der Anzahl der Städte erzeugt. Dann werden die Integer-Werte des Vektors (Chromosoms) an diesen beiden Indizees vertauscht. Dabei muss die Eigenschaft, dass Start- und Zielwert identisch sind aufrecht erhalten bleiben.
Beispiel:
Wenn für die Mutation des Chromosoms
- [ 7 8 10 2 1 3 6 5 9 4 7 ]
das zufällige Indexpaar gleich (3,5) ist, dann werden der 3. und der 3. Wert im Chromosom vertauscht. Der neue Vektor nach der Mutation ist dann
- [ 7 8 10 3 1 2 6 5 9 4 7 ]
2.3.6. Austausch¶
In jeder Iteration werden in der Population die beiden schlechtesten Chromosomen, d.h. diejenigen mit den geringsten Fitness-Werten, durch die beiden neuen Chromosomen ersetzt, falls letztere bessere Fitness-Werte haben. Dieser Austausch findet jedoch nur dann statt, wenn das neue Chromosom nicht schon in der alten Population vorhanden war. Damit wird gewährleistet, dass in der Population alle Chromosomen verschieden sind.
2.4. Realisierung mit Python und Numpy¶
Der unten aufgeführte Algorithms erzeugt einen Plot der Form:
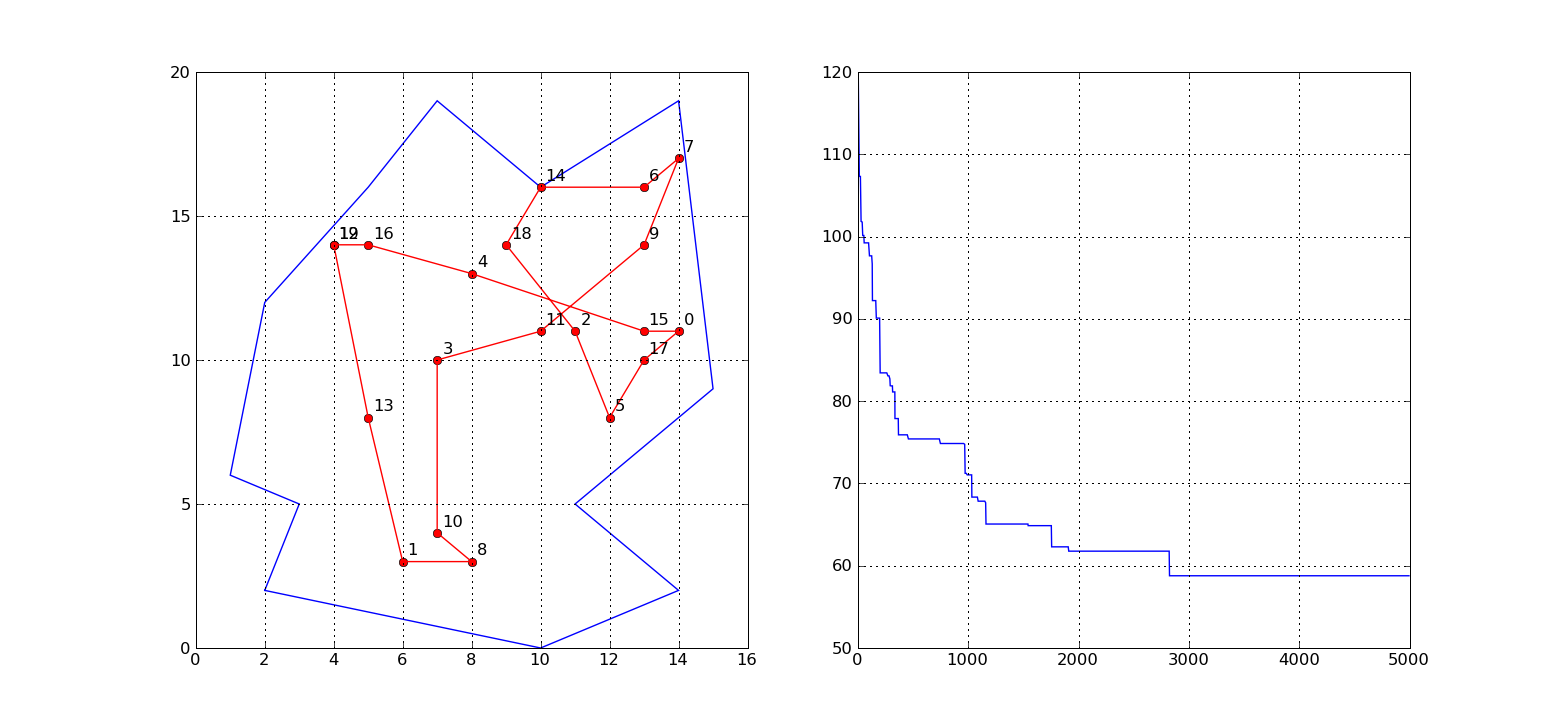
In der rechten Abbildung ist die Abnahme der Kosten (Pfaddistanz) über die insgesamt 5000 Iterationen zu sehen.
# -*- coding:utf-8 -*-
import numpy as np
import matplotlib.pyplot as plt
from matplotlib.nxutils import pnpoly
from scipy.spatial.distance import pdist,squareform
#Definition von Konstanten für die Anzeige der Stadtindizess
xshift=0.2
yshift=0.2
#Anzahl der Städte
cities = 20
#X- und Y- Koordinaten des Polygons, innerhalb dessen die Städte liegen
x=np.array([2, 10, 14, 11, 15, 14, 10, 7, 5, 2, 1, 3, 2])
y=np.array([2, 0, 2, 5, 9, 19, 16, 19, 16, 12, 6, 5, 2])
Verts=np.array([x,y]).transpose()
#Erzeugen der Städte und der Plot der Städte in der grafischen Oberfläche
plt.figure(1)
plt.subplot(121)
plt.plot(x,y)
plt.grid(True)
plt.hold(True)
i=0
np.random.seed(seed=346466)
locations=np.zeros((cities,2)) #Zufällige Festlegung der Orte
while i in range(cities):
xp=np.random.randint(0,max(x))
yp=np.random.randint(0,max(y))
isin=pnpoly(xp,yp,Verts) #testet ob der Punkt innerhalb des Polygons liegt
print xp , yp, isin
if isin:
locations[i,:]=[xp,yp]
plt.plot([xp],[yp],'ro')
plt.text(xp+xshift,yp+yshift,str(i))
i+=1
print locations
#Berechnung der euklidischen Distanz zwischen allen möglichen Stadtpaaren
distances = squareform(pdist(locations,'euclidean'))
print distances
np.random.seed()
###########################################################################
# Genetischer Algorithmus zur Lösung des TSP #
###########################################################################
#Definition der Konstanten für den GA
ITERATIONS=5000;
POPSIZE=16;
CROSSPROP=0.99;
MUTPROP=0.05;
bestDist=np.zeros(ITERATIONS) #In diesem Array wird für jede Iteration die beste Distanz gespeichert
#Erzeugen einer zufälligen Startpopulation
population=np.zeros((POPSIZE,cities+1))
for j in range(POPSIZE):
population[j,0:cities]=np.random.permutation(cities)
population[j,cities]=population[j,0]
print population
cost=np.zeros(POPSIZE)#Speichert die Kosten jedes Chromosoms der aktuellen Population
#Berechnung der Kosten jedes Chromosoms
##################################################################################################
for it in range(ITERATIONS):
#1.Berechne Fitness der aktuellen Chromosomen#################################################
for j,pop in enumerate(population):
cost[j]=0
for z in range(cities):
cost[j]=cost[j]+distances[pop[z],pop[z+1]]
sortedIndex=cost.argsort(axis=0)#Indizees der nach ansteigenden Kosten sortierten Chromosomen
sortedCost=cost[sortedIndex] #die ansteigend sortierten Kosten
bestDist[it]=sortedCost[0]
sortedPopulation=population[sortedIndex] #Sortierung der Population nach ansteigenden Kosten
InvertedCost=1/sortedCost #Berechung des Nutzen (Fitness) aus den Kosten
#InvertedCost enthält die berechneten Fitness-Werte
if it%100==0:
print '-'*10+' Iteration: ',it
print InvertedCost[0]
print sortedPopulation[0]
#2.Selektion: Zufällige Auswahl von Chromosomen aus der Population####################
#Mit dem folgenden Prozess wird gewährleistet, dass die Wahrscheinlichkeit für die
#Selektion eines Chromosoms umso größer ist, je größer sein Nutzenwert ist.
InvertedCostSum = InvertedCost.sum()
rn1=InvertedCostSum*np.random.rand()
found1 = False
index=1
while not found1:
if rn1<InvertedCost[:index].sum(axis=0):
found1=index
else:
index+=1
found1=found1-1
equal=True
while equal:
rn2=InvertedCostSum*np.random.rand()
found2 = False
index=1
while not found2:
if rn2<InvertedCost[:index].sum(axis=0):
found2=index
else:
index+=1
found2=found2-1
if found2 != found1:
equal=False
parent1=sortedPopulation[found1]
parent2=sortedPopulation[found2]
########## parent1 und parent2 sind die selektierten Chromsomen##############################
#3.Kreuzung####################################################################################
crossrn=np.random.rand()
if crossrn<CROSSPROP:
cp=np.ceil(np.random.rand()*cities)
head1=parent1[:cp]
tailind=0
tail1=np.zeros(cities-cp+1)
for a in range(cities):
if parent2[a] not in head1:
tail1[tailind]=parent2[a]
tailind+=1
tail1[-1]=head1[0]
head2=parent2[:cp]
tailind=0
tail2=np.zeros(cities-cp+1)
for a in range(cities):
if parent1[a] not in head2:
tail2[tailind]=parent1[a]
tailind+=1
tail2[-1]=head2[0]
child1=np.append(head1,tail1)
child2=np.append(head2,tail2)
#child1 und child2 sind die Ergebnisse der Kreuzung###############################################
#4. Mutation#########################################################################################
mutrn=np.random.rand()
if mutrn<MUTPROP:
mutInd=np.ceil(np.random.rand(2)*(cities-1))
first=child1[mutInd[0]]
second=child1[mutInd[1]]
child1[mutInd[0]]=second
child1[mutInd[1]]=first
child1[-1]=child1[0]
mutrn=np.random.rand()
if mutrn<MUTPROP:
mutInd=np.ceil(np.random.rand(2)*(cities-1))
first=child2[mutInd[0]]
second=child2[mutInd[1]]
child2[mutInd[0]]=second
child2[mutInd[1]]=first
child2[-1]=child2[0]
#child1 und child2 sind die Resultate der Mutation################################################
#5. Ersetze die bisher schlechtesten Chromosomen durch die neu gebildeten Chromosomen, falls die neuen
#besser sind
costChild1=0
costChild2=0
for z in range(cities):
costChild1=costChild1+distances[child1[z],child1[z+1]]
costChild2=costChild2+distances[child2[z],child2[z+1]]
replace1=False
replace2=False
index=POPSIZE-1
while index > 0:
if sortedCost[index]>costChild1 and not replace1:
if not np.ndarray.any(np.ndarray.all(child1==sortedPopulation,axis=1)):
sortedPopulation[index]=child1
replace1=True
elif sortedCost[index]>costChild2 and not replace2:
if not np.ndarray.any(np.ndarray.all(child2==sortedPopulation,axis=1)):
sortedPopulation[index]=child2
replace2=True
if replace1 and replace2:
break
index=index-1
population=sortedPopulation
######################################Ende der Iteration#############################
#Graphische Anzeige der Kosten über die Iterationen und graphische Anzeige des gefundenen Weges
xcoords=[]
ycoords=[]
for i in range(cities+1):
xcoords.append(locations[sortedPopulation[0,i],0])
ycoords.append(locations[sortedPopulation[0,i],1])
plt.plot(xcoords,ycoords,'r-')
print "Best distance ",bestDist[-1]
print "Population ",population[0]
plt.subplot(122)
plt.grid(True)
plt.plot(range(ITERATIONS),bestDist)
plt.show()